QPix 미생물 콜로니 피커
시간당 최대 3000개의 콜로니를 픽업할 수 있는 자동화된 미생물 스크리닝 시스템
시간당 최대 3,000개의 콜로니를 픽업할 수 있는 자동화된 미생물 스크리닝 시스템
QPix® 미생물 콜로니 피커는 동급 최강의 콜로니 피킹 기술을 활용하여 병목 현상을 완화하고 방대한 유전자 라이브러리를 빠르고 정확하고 효율적으로 Screening합니다. 사용하기 쉬우며 직관적인 소프트웨어는 사용자가 정밀한 로봇으로 매번 알맞은 콜로니를 피킹할 수 있는 콜로니 피킹 실행을.. 미생물 스크리닝 이외에도, 이 시스템은 박테리아 액상 배양 및 한천배지에서의 플레이팅 등 여러 샘플 준비 및 플레이트 취급 과정을 자동화합니다.
데이터는 자동으로 기계의 데이터베이스에 기록되어 사용자에게 완전한 감사 추적과 샘플 추적을 제공하여 데이터가 손실되지 않도록 합니다. 확장 가능한 모듈식 콜로니 피커 시리즈를 사용하면 모든 크기의 샘플에 대한 실험과정의 정확도와 처리량을 높이는 동시에 추가적인 처리량 증가가 가능합니다.

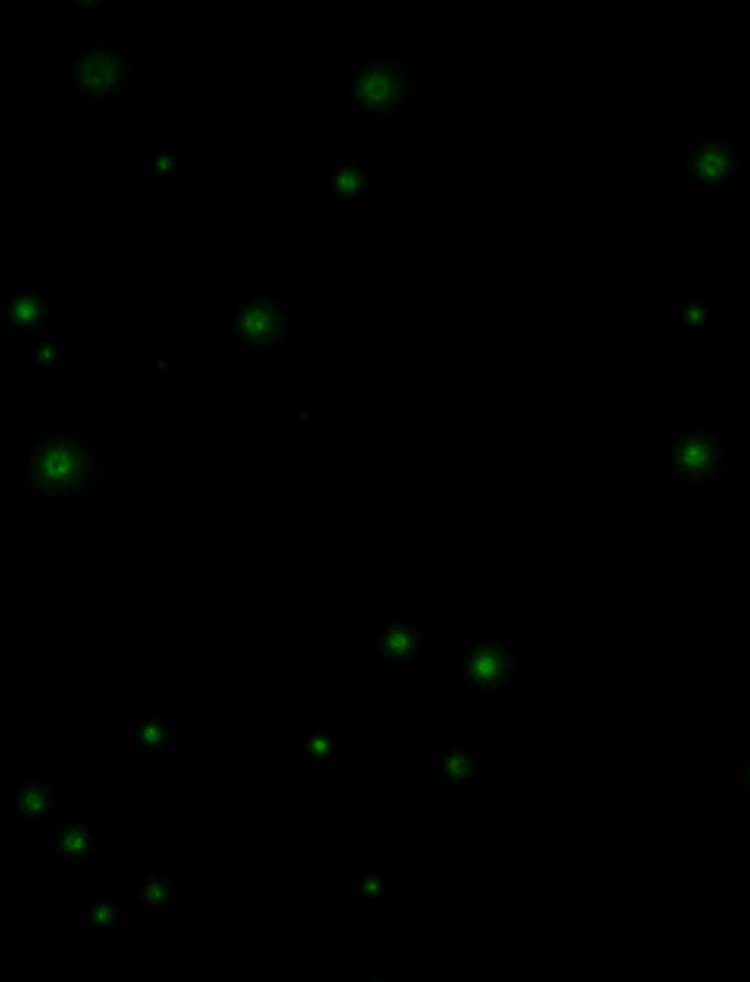
원하는 표현형을 이용한 콜로니 식별
QPix 콜로니 피커는 다양한 종류의 미생물 및 형광의 세기, 청색/백색 선택, 크기 및 근접성, 억제영역을 포함한 다중 선택 양식을 지원합니다.

효율적인 콜로니 선택
유기체 특이적 핀과 한천배지 센서 제품군은 효율적인 피킹을 보장합니다. 이 시스템은 98%를 초과하는 피킹 효율성을 제공하여 계속 대기하지 않아도 안심할 수 있도록 합니다.

멸균 유지관리
기기 내부의 위생처리, 핀 세척, 핀의 할로겐 건조를 위한 UV 광선을 포함한 멸균 기능을 사용할 수 있습니다.
기능

유기체 특이적 핀
서로 다른 모양과 피킹 영역 핀으로 E. coli, 파지, 효모에 대한 효율성을 극대화합니다. 플레이팅 특이적 핀은 한천배지에 액체 배양액이 균일하게 분포하도록 합니다.

다양한 이미징 모드
Colony는 백색광, 형광, 색을 이용하여 사전에 지정된 Parameter를 기반으로 피킹할 수 있습니다. 필터의 사용은 청색-백색 Colony 스크리닝과 같은 응용 작업이 가능하게 합니다.

플레이팅과 스프레딩
샘플 96개의 자동화 플레이팅 및 선상도말법은 30분 이내에 완료되므로, 대기 시간을 줄여줍니다.

복제, 그리드 및 유효물질 피킹
자동화 플레이트 취급 및 추적으로 다운스트림 assay와 샘플 관리를 간소화할 수 있습니다. QPix 콜로니 피커는 유연한 플레이트 복제, 그리딩, 유효물질 피킹 기능을 제공합니다.

한천배지 감지
한천배지의 초음파 높이 센서는 최대 피킹 효율에 도달하도록 하는 다양한 주입량으로 인한 높이의 차이를 감지합니다.

규모 조절이 가능한 자동화 옵션*
QPix HT 모델은 모듈식 덱이 있는 로봇 호환 솔루션입니다. 고급 워크플로 엔지니어링 솔루션 팀은 다양한 고객 서비스를 통해 콜로니 피커를 맞춤화할 수 있습니다.
*가격, 배송 기간, 제품 사양은 상호 간에 동의한 기술적 요구 사항에 따라 달라집니다. 솔루션 요구 사항으로 인해 표준 성능이 조정될 수 있습니다.
QPix 콜로니 피커를 사용한 실험과정의 자동화
콜로니 피킹은 생물학적 연구에서 필수적인 단계입니다. 과학자들은 DNA 또는 단백질을 대량 생산하여 다양한 응용 분야의 다운스트림에 사용하기 위해 미생물 클론을 종종 분리합니다. 일반적으로, 콜로니 피킹은 멸균 상태의 피펫 팁, 접종 루프를 사용하여 수동으로 실시하며, 보통 느리고, 노동집약적이며 시간이 많이 듭니다. 자동화 콜로니 피커는 전체 과정을 더 빠르게 만들 뿐만 아니라, 결과 또한 더 일관적이고 신뢰할 수 있습니다.
어떤 QPix가 여러분에게 적합한가요?
imaging
picking-capacity
colony-selection-criteria
picking-and-regional-picking
barcode-tracking
re-arraying-and-replication
gridding
plating-and-streaking
agar-to-agar
robotics-integration
shaking-incubator
liquid-handler
pcr
sealer-peeler
elisa
destination-plate-capacity
stackers
source-plate-capacity
walkaway-time
*가격, 배송 기간, 사양은 상호간에 동의한 기술적 요구 사항에 따라 달라집니다. 솔루션 요구 사항으로 인해 표준 성능이 조정될 수 있습니다.
맞춤형 솔루션에는 https://moleculardevices.com/custom-products-purchase-terms에서 이용 가능한 Molecular Devices의 맞춤형 제품 구매 약관이 적용됩니다.
자주 묻는 질문(FAQ)
QPix 미생물 콜로니 피커란?
QPix 미생물 콜로니 피커는 High-Throughput 미생물 콜로니 선택과 피킹을 위해 설계된 고급 자동화 시스템입니다. 이는 클론 스크리닝 과정을 간소화하고 수동 콜로니 피킹을 할 필요가 없도록 합니다.
QPix 미생물 콜로니 피커는 어떻게 작동하나요?
QPix 미생물 콜로니 피커는 Imaging과 로봇 기술의 조합을 활용하여 한천 플레이트에서 개별 미생물 콜로니를 정확하게 식별하고 피킹합니다. 정교한 알고리즘을 사용하여 콜로니 크기, 모양, 기타 특성을 분석하여 정확한 Colony Selection이 가능합니다.
QPix 미생물 콜로니 피커의 주요 기능은 무엇인가요?
QPix 미생물 콜로니 피커는 High-Resolution Imaging, 사용자 정의 가능한 Colony Selection 기준, 자동화된 피킹과 재배열, 조정 가능한 피킹 Parameter, 콜로니 추적과 데이터 분석을 위한 직관적인 소프트웨어를 포함한 여러 주요 기능을 제공합니다.
QPix 미생물 콜로니 피커를 사용하면 어떤 이점이 있나요?
QPix 미생물 콜로니 피커는 콜로니 피킹 과정을 자동화하여 클론 스크리닝의 처리량과 효율성을 크게 높입니다. 교차 오염 위험을 최소화하고 정확성과 재현성을 향상하며 연구자가 귀중한 시간과 자원을 확보합니다.
QPix 미생물 콜로니 피커의 이점을 누릴 수 있는 응용 분야는 무엇인가요?
QPix 미생물 콜로니 피커는 재조합 단백질 발현, 항체 개발, 합성 생물학, 방향성 진화, 균주 공학을 포함한 광범위한 응용 분야에 이상적입니다. 학술 연구, 제약 개발, 생명공학, 효율적인 클론 스크리닝이 필요한 기타 산업에 적합합니다.
QPix 미생물 콜로니 피커의 주요 기능은 무엇인가요?
QPix 미생물 콜로니 피커는 자동화된 콜로니 피킹, 식별, 추적 기능을 제공합니다. 이를 통해 연구자는 한천 플레이트에서 개별 미생물 콜로니를 효율적으로 선택하고 옮길 수 있어 시간을 절약하고 인적 오류를 줄일 수 있습니다.
QPix 미생물 콜로니 피커는 어떤 실험과정에서 사용할 수 있나요?
QPix 미생물 콜로니 피커는 다양한 실험과정에 통합될 수 있는 다용도 도구입니다. 이는 클론 스크리닝, 돌연변이 라이브러리 생성, 미생물 유전체학, 방향성 진화, High-Throughput 균주 공학과 같은 응용 분야에서 일반적으로 사용됩니다.
QPix 미생물 콜로니 피커는 어떻게 과학 연구를 최적화하나요?
QPix 미생물 콜로니 피커는 클론 스크리닝과 콜로니 피킹의 과정을 간소화하여 연구자가 더 짧은 시간에 더 많은 수의 콜로니를 분석할 수 있도록 합니다. 이러한 최적화는 연구 프로젝트의 효율성을 향상시키고 개발을 가속화하며 다운스트림 분석과 실험을 촉진합니다.
QPix 미생물 콜로니 피커는 다양한 유형의 한천 플레이트를 처리할 수 있나요?
예, QPix 미생물 콜로니 피커는 페트리 접시, 다중 Well 플레이트, 기타 맞춤형 플레이트를 포함한 다양한 한천 플레이트 형식을 처리하도록 설계되었습니다. 이 시스템은 유연하며 다양한 실험의 특정 요구 사항에 적응할 수 있습니다.
QPix 미생물 콜로니 피커와 함께 제공되는 소프트웨어는 어떻게 연구 생산성을 향상시키나요?
QPix 미생물 콜로니 피커와 함께 제공되는 소프트웨어는 실험 설계, Colony Selection 기준 사용자 정의, 데이터 분석을 위한 직관적인 사용자 인터페이스를 제공합니다. 이를 통해 콜로니를 효율적으로 추적하고 실험과정 관리를 용이하게 하며 데이터 기반 의사 결정을 위한 통찰력 있는 지표를 제공합니다.
최신 자료
제품 응용 분야
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/biofuels
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/blue-white-screening
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/dna-sequencing
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/monoclonal-antibodies-mabs
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/phage-display
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/protein-evolution
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/synthetic-biology
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/plasmid-production
고객 사례 소개

당신의 다음 연구와 새로운 발견을 위해
어떤 도움이 필요하신가요?
뛰어난 역량의 직원들이 고객분들의 연구과제의 문제 해결을 위해 일선에서 대기하고 있습니다.
원격 또는 현장 제품 데모, 웨비나 등을 통해 도움을 받으실 수 있습니다. 무엇을 도와 드릴까요?
아래 항목 중에서 선택하십시오.